Variabilità del Cipresso comune nel Trentino-Alto Adige
di Nicola La Porta

Cipresso comune – Cupressus sempervirens L. (foto Nicola La Porta)
INTRODUZIONE
Il cipresso comune (Cupressus sempervirens L.) è una pianta originaria delle regioni più orientali del bacino mediterraneo: Turchia e isole greche. La sua introduzione in Italia risale probabilmente al periodo etrusco. Si è poi diffuso in tutto il mediterraneo adattandosi ottimamente alle nostre condizioni ambientali, tanto da essere considerato una specie naturalizzata.
Il Cipresso, oltre ad avere un ruolo importante nella caratterizzazione del paesaggio mediterraneo per la sua funzione estetica, ha dimostrato di essere una specie pioniera insostituibile per il rimboschimento dei terreni rocciosi, argillosi, calcarei, aridi e superficiali. Esercita una funzione di prevenzione dall’ erosione idrogeologica e contemporaneamente costituisce una fonte di reddito per l’ottima qualità del legno che produce. Notevole anche la sua utilizzazione come pianta frangivento.
In Italia non sono presenti boschi naturali di C. sempervirens. Cipressete di piccole dimensioni sono reperibili nelle colline costiere dalla Liguria alla Calabria in Sicilia. Nell’Italia centrale, particolarmente in Toscana, si trovano le cipressete più consistenti e produttive. Questa specie è presente anche a nord degli Appennini in boschetti, alberature e piante ornamentali localizzate principalmente intorno ai grandi laghi alpini (Xenopoulos,1990).
Negli ultimi 50 anni è stato attaccato da un fungo parassita, il Seiridium cardinale, comunemente conosciuto con il nome di “cancro del Cipresso”, il quale ha messo in serio pericolo la sopravvivenza di questa arborea in tutta Italia e in altri paesi mediterranei.
Nella regione Trentino-Alto Adige il cipresso è importante esclusivamente per gli aspetti estetici e paesaggistici. La diffusione crescente di questa pianta arborea sul territorio regionale e la possibilità che in futuro i cambiamenti del clima rendano l’area in oggetto ancora più favorevole alla coltivazione, hanno suggerito di prendere in considerazione uno studio di questa conifera in Trentino-Alto Adige.
In regione il cipresso si trova spesso a vegetare in condizioni difficili soprattutto per i ricorrenti danni da basse temperature (Raddi, 1989). Benché questi danni generalmente non siano la causa diretta del deperimento della pianta agiscono spesso indirettamente nell’aumentare le capacità di penetrazione delle spore del fungo (Manion, 1991). In questo contesto, piante più resistenti a danni da freddo, oltre ad adattarsi meglio ad un territorio con più frequenti eventi di minimi termici, garantiscono maggiori difese anche contro attacchi di S. cardinale (Intini, 1976).
La Provincia Autonoma di Trento ha pertanto finanziato un progetto denominato Ecocypre mirato alla “Valutazione ecologica e gestione sostenibile del cipresso nel paesaggio del Trentino”allo scopo di mettere a disposizione della comunità materiale genetico selezionato per la resistenza alle avversità biotiche (Seiridium cardinale) e abiotiche (minimi termici).
Presso lo IASMA (Istituto Agrario di S. Michele a/A) viene valutata la variabilità genetica delle popolazioni di cipresso diffuse nel Trentino-Alto Adige e delle popolazioni del suo patogeno S. cardinale ai fini della loro caratterizzazione e valutazione della biodiversità.
Contemporaneamente viene portato avanti uno screening precoce di 100 cloni di C. sempervirens (selezionati in precedenti programmi di ricerca per la resistenza al cancro) per l’adattamento alle condizioni climatiche del Trentino-Alto Adige (tolleranza a basse temperature).
Lo scopo di questa ricerca è valutare la variabilità genetica delle popolazioni dell’ospite nella regione per stimare l’impatto ambientale che potrebbe avere l’eventuale introduzione nel territorio di nuovi cloni selezionati resistenti al Seiridium cardinale e in grado di sopravvivere ai rigidi inverni trentini.
Inoltre è sicuramente interessante caratterizzare le popolazioni che vivono al limite settentrionale dell’area di diffusione del cipresso anche nell’ottica di utilizzare i dati ottenuti per un confronto con materiale proveniente da altre regioni italiane e mediterranee.

Cipressi nei pressi di Arco di Trento (foto Nicola La Porta)
MATERIALI E METODI
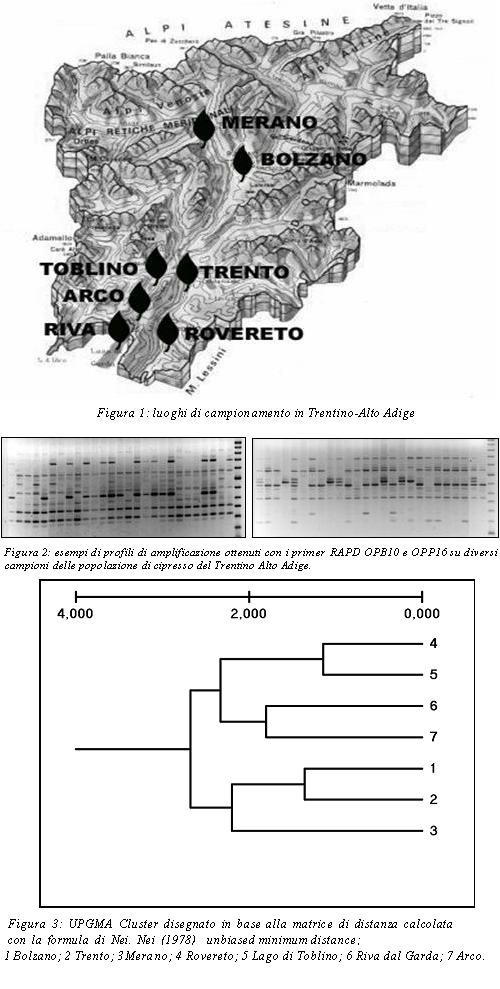
Materiale vegetale- Lo studio di variabilità genetica è stato condotto su sette popolazioni di Cupressus sempervirens L. (Cupressaceae) provenienti da diverse zone del Trentino-Alto Adige (figura 1) rappresentative di buona parte delle aree di diffusione del cipresso nella regione. Il diametro del fusto di ciascuna pianta campionata è superiore ai trenta centimetri allo scopo di valutare la biodiversità delle piante più antiche e ben adattate alle condizioni climatiche della regione.
Estrazione del DNA– Circa 100 mg di foglie di ciascun campione sono state liofilizzate con una pompa a vuoto nell’arco di 36 ore. Il materiale fogliare liofilizzato e congelato in azoto liquido è stato polverizzato sfruttando il “Mixer Mill MM 300” (Qiagen GmbH, Hilden, Germania). I successivi passaggi di estrazione sono stati eseguiti con il Kit della Qiagen “Dneasy plant mini kit” seguendo il protocollo.
Per consentire una valutazione visiva della quantità e della qualità del DNA, un microlitro di ciascun prodotto di estrazione è stato caricato su gel di agarosio all’1% in buffer TBE 0,5X, postcolorato con etidio bromuro e fotografato al transilluminatore. Ciascun campione è stato poi confrontato visivamente con uno standard l a concentrazione nota di DNA caricato sul gel.
Amplificazione RAPD– Non essendo ancora presenti in letteratura marcatori molecolari specifici per il cipresso, abbiamo utilizzato la tecnica RAPD in quanto non richiede la conoscenza di sequenze geniche a priori per potere effettuare analisi molecolari di variabilità.
64 primer RAPD 10mer della Operon Primer Technology (kit OPB, kit OPP e kit OPE e OPF3 OPH7 OPL6 e OPL19) sono stati testati su DNA di Cupressus sempervirens con PCR “gradient” (gradiente da 36 a 48°C) per valutare quali primer fossero in grado di produrre il maggior numero di amplificati risolvibili su gel di agarosio e quale fosse la temperatura di annealing ottimale per ciascuno d essi.
I 23 primer che hanno dato risultati soddisfacenti in questa prima prova sono stati testati con HotStarTaq Polimerase (Qiagen) per mettere a punto le condizioni di amplificazione (T annealing, MgCl2, numero cicli di amplificazione) e per verificare il polimorfismo e la riproducibilità dei profili di amplificazione.
Nove primer sono stati infine selezionati per i successivi studi sui 210 genotipi di cipresso (omissis).
Analisi statistiche– I frammenti amplificati, identificati con il nome del primer usato e con la massa molecolare espressa in paia di basi, sono stati rilevati manualmente e tradotti in matrici numeriche di presenza assenza (1 presenza e 0 assenza) che riproducono i differenti fenotipi RAPD caratterizzati.
Per l’analisi statistica dei dati sono stati utilizzati tre programmi: POGENE32 (Francis, 1999) TFPGA (Miller, 1997) e ARLEQUIN (Schneider, 2000)
POPGENE 32 è stato usato per valutare la diversità genetica all’interno delle popolazioni (HS), la diversità genetica totale (HT) e il coefficiente di differenziazione (Gst), per calcolare l’indice di diversità di Shannon (Shannon, 1949) e l’indice di diversità di Nei (1973), il numero e la percentuale di marcatori polimorfici e monomorfci, la media del numero di alleli osservati (na) e il numero effettivo degli alleli (ne) (Kimura and Crow; 1964).
TFPGA è stato usato per calcolare le matrici di distanza tra le popolazioni (Nei’ s unbiased genetic distance; Nei, 1978) e per disegnare il relativo dendrogramma con il metodo UPGMA.
ARLEQUIN e stato usato per valutare l’unicità di ciascun fenotipo amplificato, per le analisi della varianza molecolare (AMOVA), per calcolare una matrice di distanza basata sui valori della Fst e la loro significatività.
RISULTATI
Fingerprinting delle popolazioni di Cupressus sempervirens–
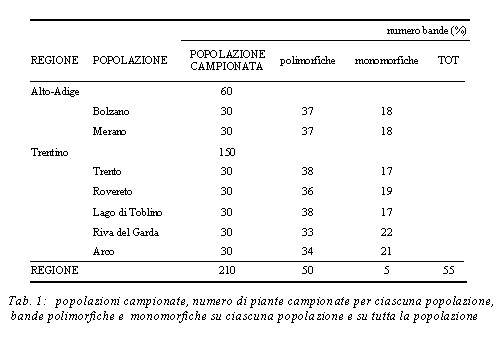
I nove primer selezionati hanno amplificato 55 bande (con un media di 6,1 bande per primer) di dimensioni variabili da un minimo di 350 bp a un massimo di 1420 bp 50 bande (90,91%) su 55 sono polimorfiche su tutta la popolazione regionale, 5 (9,09%) sono monomorfiche e 47 (85,45%) seguono il criterio di Linch e Milligan (Tab. 1).
La percentuale di bande polimorfiche all’interno di ciascuna popolazione varia dal 60% di Riva del Garda al 69.09% del Lago di Toblino e di Trento.
In figura 2 sono riportati i profili di amplificazione dei primer OPP16 e OPB10 su diversi campioni di cipresso che mostrano chiaramente il polimorfismo presente all’interno delle popolazioni.
Variabilità genetica intrapopolazione–
La partizione della variabilità genetica intrapopolazione è stata calcolata con diversi indici su tutti i 55 marcatori selezionati.
Il numero medio di alleli na varia da 1,600 a 1,691 (popolazione di Trento). Il numero di alleli effettivo ne, che può essere usato come misura della variabilità genetica di una popolazione o di una specie, varia da 1,317 a 1,387 (Trento).
I valori dell’ indice di diversità di Nei (1973) calcolati su ciascuna popolazione variano da un minimo di 0,196 a un massimo di 0,229 (Trento) in accordo con l’indice di diversità di Shannon che assume un valore minimo di 0,301 e un valore massimo 0,346 (Trento).
L’indice di diversità di Nei (h) calcolato su tutta la popolazione ha un valore di 0.230. L’indice di diversità di Shannon è 0,358.
La popolazione di Trento mostra in tutti i casi la variabilità intrapopolazione più alta.
La media della variabilità genetica intrapopolazione HS è 0,209, la diversità genetica totale (HT) 0,230, il coefficiente di differenziazione tra le popolazioni (Gst) è 0,09.
Tutti i valori trovati indicano una bassa variabilità sia tra gli individui all’interno delle popolazioni sia tra le popolazioni. I dati concordano con i valori ottenuti dalle analisi della varianza molecolare.
Le analisi AMOVA indicano infatti che la maggior parte della variabilità (94,16%) è stata riscontrata tra gli individui mentre il rimanente 5,84% (Fst = 0,0584 P value< 0,001 bootstrap con 1023 ripetizioni) è dovuto alla variabilità tra le popolazioni.
Il valore della Fst è simile a quello della Gst ottenuto dall’indice di diversità di Nei.
E’ stata calcolata una matrice di distanza fra coppie di popolazioni. I valori della Fst sono significativi (P<0,05), tranne che tra le popolazioni di Rovereto e Toblino e di Trento e Bolzano.
Con TFPGA è stata calcolata una seconda matrice di distanza (Unbiased Genetic Distance; Nei 1978), significativamente correlata con la prima, sulla quale è stato disegnato un dendrogramma con il metodo UPGMA.
Non c’è correlazione tra la matrice delle distanze genetiche e la matrice delle distanze geografiche fra le popolazioni (Mantel test).
DISCUSSIONE
Fino ad oggi la variabilità genetica del Cupressus sempervirens L. è stata indagata principalmente con gli isoenzimi (Raddi S. 1999; Papageorgiou 1993; Papageorgiou 1994) su popolazioni naturali provenienti da paesi del Mediterraneo orientale (Grecia ,Turchia e Israele), zone di origine di questa conifera.
I valori di eterozigosità attesa trovati tra i loci polimorfici di queste popolazioni risultano essere simili tra loro (Turchia Hexp = 0.350, Raddi S. 1999; Grecia Hexp= 0,400 Papgeorgiu et al 1994; e Israele Hexp = 0.479, Shiller and Korol, 1997) e superiori a quelli osservati in altre conifere boreali (Hamrick et al, 1992) ed in altre Cupressaceae (Uchida et al. 1997 e Conkle,1997).
La diversità genetica del cipresso nelle sue zone di origine è quindi molto elevata e probabilmente mantenuta tale da una alta capacità di incrocio e dalla presenza di popolazioni numerose (Crow and Kimura …).
Ad oggi non sono conosciute sequenze del genoma che possano essere utilizzate per studi di variabilità genetica sul Cupressus sempervirens. La tecnica RAPD è stata scelta perché è in grado di fornire un buon numero di marcatori in breve tempo e senza la necessità di preventive informazioni sulla sequenza del DNA. Inoltre i RAPD generalmente rilevano valori di diversità genetica simili a quelli ottenuti con gli allozimi (Aagaard, 1998; Liu, 1993).
Valutando i risultati di questo lavoro appare evidente che la biodiversità della popolazione del Trentino-Alto Adige è ridotta rispetto alle potenzialità della specie. La popolazione “Trento” risulta essere la più eterogenea anche se i valori di variabilità intrapopolazione dei diversi luoghi di campionamento (indice di diversità di Nei = h e di Shannon = I) sono molto simili tra loro.
Un risultato che tuttavia non stupisce se consideriamo la superficie di campionamento ristretta e localizzata al limite nord dell’ area italiana di vegetazione del cipresso unita alla forte azione antropica che nei secoli ha influenzato gli equilibri genici di questa specie in Italia e soprattutto nel Trentino-Alto Adige.
Inoltre generalmente la biodiversità delle popolazioni che vegetano ai margini dell’area di diffusione di una specie è inferiore rispetto a quella di popolazioni che crescono più vicine alle zone di origine o in prossimità di un antico rifugio glaciale (Collignon, 2000).
I valori di diversità genetica riscontrati tra le popolazioni di cipresso del Trentino-Alto Adige (Gst = 0,09) sono dello stesso ordine di grandezza sia di quelli trovati fra le popolazioni naturali della Turchia (Gst = 0,073, Raddi S.1999), sia di quelli riscontrati tra le conifere europee (Gst = 0,068, Hamrick et al 1989; Picea Abies popolazioni italiane Gst= 0,044, Giannini, 1991), sia dei valori di altre piante arboree che nel corso dei secoli hanno subito l’ influenza antropica (Q. rubur Gst nelle popolazioni Europee varia da un massimo di 0,23 in Polonia a un minimo di 0,014 in Romania Yakovlev, 2002).
Le distanze genetiche trovate tra le popolazioni sono basse ma significative anche se sono solo parzialmente ordinate in accordo con la collocazione geografica (figura 3). Le più simili sono “Trento” con “Bolzano” e “Rovereto” con “Toblino”. Probabilmente le piante che popolano la regione provengono da diverse località italiane e sono state importate nelle differenti zone di campionamento con eventi indipendenti, tali da conservare una discreta diversità tra le diverse popolazioni.
La bassa variabilità genetica totale (Ht = 0,23) e intrapopolazione (Hs = 0,209) può essere spiegata con la selezione morfologica che le piante hanno probabilmente subito ad opera dell’uomo: la maggior parte dei cipressi in regione presenta infatti forma della chioma fastigiata o intermedia considerate di maggior pregio estetico. Non si possono invece fare speculazioni sulla selezione dovuta alle condizioni climatiche perché non ci sono ancora dati sufficienti sulla variabilità di altre popolazioni italiane e del bacino mediterraneo.
Lo scopo di questo lavoro è stimare l’impatto ambientale che potrebbe avere l’eventuale introduzione nel territorio di cloni selezionati nell’ambito del progetto Ecocypre tolleranti alle basse temperature e resistenti al Seiridium cardinale. Dai dati ottenuti fino ad ora si può pensare che l’introduzione di cloni non comporterebbe alterazioni significative degli equilibri genici e della variabilità già bassa della popolazione locale.
BIBLIOGRAFIA
– AAGAARD J.E., KONSTANTIN V. K., STEVEN H.S., 1998. RAPDs and allozymes exibit similar levels of diversity and differentiation among populations and races of Douglas-fir. Heredity 81:68-78.
– COLLIGNON A.M., FAVRE J.M.,2000. Contribution to the postglacial history at the western margin of Picea abies natural area using RAPD markers. Annals of Botany 85:713-722.
– CONKLE M.T., 1987. Electrophoretic analysis of variation in native Monterey cypress (Cupressus macrocarpa Hartw.). In: Elias T.S. (ed.), Conservation and menagement of rare and Endangered Plants. Conf. Proc. Sacramento,CA, USA, 249-256.
– CROW J.F., KIMURA M.,1970. An introduction to population genetic theory. Harper & Row, New York.
– FRANCIS C.Y., RONG-CAI Y. (University of Alberta), BOYLE T. (Centre for International Forestry Research), 1999. POPGENE version 1.32, Microsoft Window-based Freeware for Population Genetic Analysis.
– GIANNINI R., MORGANTE M., VENDRAMIN G.G., 1991. Allozyme variation in Italian population of Picea Abies (L.) Karst. Silvae Genetica, 40:160-166.
– HAMRICK J.L., GODT M.J.W., 1989. Allozyme diversity in plant species. In: Brown A.D.H., Clegg M.T., Kahler A.L. Wier, B.S. (Eds.), Plant Population Genetics, Breeding and Genetics Resources. Sinauer Associates, Inc, Sunderland, MA: 43-63.
– HAMRICK J.L., GODT M.J.W.,SHERMAN-BROYLES S.L., 1992. Factors influencing levels of genetic diversity woody plant species. In: Adams W.T., Strauss S.H., Copes D.L., Griffin A.R., Population genetics of forest Trees. Kluwer Academic Publishers, Dordrecht, pp 95-124.
– INTINI M., PANCONESI A., 1976. Alcuni aspetti della biologia del Coryneum cardinale Wag. in Toscana. Ann. Accad. It. Sci. For., 25: 1-25.
– LIU Z.,FURNIER G.R., 1993.Comparison of allozyme,RFLP, and RAPD markers for revealing genetic variation within and between trembling aspen and bigtooth aspen.Theor.Appl. Genet., 87:97-105
– MANION P.D., 1991. Tree disease concepts. Prentice-Hall, Englewood Cliffs.
– MILLER M P., 1997. TFPGA ver. 1.3, Tools for Population Genetic Analyses. Departement of Biological Scinces Northern Arizona University, Flagstaff, Arizona.
– NEI M., 1973. Analysis of gene diversity in subdivided populations. Proc. Nat. Acad. Sci. USA 70: 3321-3323.
– NEI M., (1978). Estimation of average heterozygosity and genetic distance from a small number of individuals. Genetics 89: 583-590.
– PAPAGEORGIU A.C., PANETSOS K.P., HATTEMER H.H., 1994. Genetic differentiation of natural Mediterranen cypress (Cupressus sempervirens L.) populations in Greece. For Genet. 1:1-12.
– RADDI P., PANCONESI A., 1989. Genetic variability of tolerance to cold in Cupressus sempervirens progenies. Silvae Genetica, 38: 168-172.
– RADDI S., SABRI S., 1999. Genetic diversity in Natural Cupressus sempervirens L. populations in Turkey. Biochemical Systematics and Ecology 27: 799-814.
– SHNEIDER S., ROESSLI D., EXCOFFIER L. (2000). A software for population genetic data analysis. Genetic and Biometry Laboratory, University of Geneva, Switzerland.
– SHANNON C.E., WEAVER W., 1949. The mathematical theory of communication. Univ. Of Illinois Press, Urbana.
– SHILLER G., COROL L., 1997. Electrophoresis analysis of diversity within Cupressus sempervirens L. growing in Israel. Israel Journal of Plant Science, 45: 1-8.
– UCHIDA K., TOMARU C., YAMAMOTO C. OHBA K., 1997. Allozyme variation in natural populations of Hinoki, Chamaecyparis obtusa(sieb et Zucc.) Endl. and its comparison with plus trees selected from artificial stands. Breeding Science, 47:7-14.
– WILLIAMS J.G.K., KUBELIK A.R., KENNETH J., RAFALSKY J.A., TINGEY S.V., 1990. DNA polymorphisms amplified by arbitrary primers are useful as genetic markers. Nucleic Acids Research 18: 6531 – 6535.
– XENOPOULOS S., ANDREOLI C., PANCONESI A., PINTO GANHAO J., THUSET J.J., 1990. Importance of cypress. In: ‘Progress in EEC research on cypress diseases’. Report EUR 12493 EN, 1-13.
– YAKOVLEV I.A.,KLEINSCHMIDT J., 2002. Genetic differentiation of peduncolate oak Quercus Rubur L. in the European part of Russia based on RAPD markers. Russian Journal of Genetics, 38 (2): 207-215.
ALLEGATI
Nicola La Porta, laureato in Scienze forestali presso la Facoltà di Agraria di Firenze, è ricercatore presso il Dipartimento Risorse Naturali dell’Istituto Agrario di S.Michele all’Adige (TN). Curriculum vitae >>>
 |
Atlante di selvicoltura Una vera enciclopedia sulla selvicoltura. I termini discussi nel contesto di altre voci ed i sinonimi richiamati sono più di 1500. Le specie vegetali ed animali trattate più o meno diffusamente sono più di 800. Acquista online >>> |